

分子动力学(Molecular Dynamics,MD)模拟是一套结合物理,数学和化学的综合分子模拟方法,该方法主要是依靠牛顿力学来模拟分子体系的运动,在由分子体系的不同状态构成的系综中抽取样本,计算体系的热力学量和其他宏观性质。分子动力学法是一种计算机模拟实验方法,是研究凝聚态系统的有力工具。通过分子动力学模拟,研究者得到体系原子的运动轨迹,可观察到原子运动过程的各种微观细节。通过对研究体系的动态模拟,我们能够在分子水平上理解生物大分子的运动与生物功能,蛋白-小分子之间相互作用机理,纳米材料分子的自组装过程。分子动力学模拟是理论计算和实验方法的有力补充,广泛应用于物理,化学,材料科学,生物医药等科学和技术领域。
研究方向:生物,生化,高分子,食品,材料,环境等
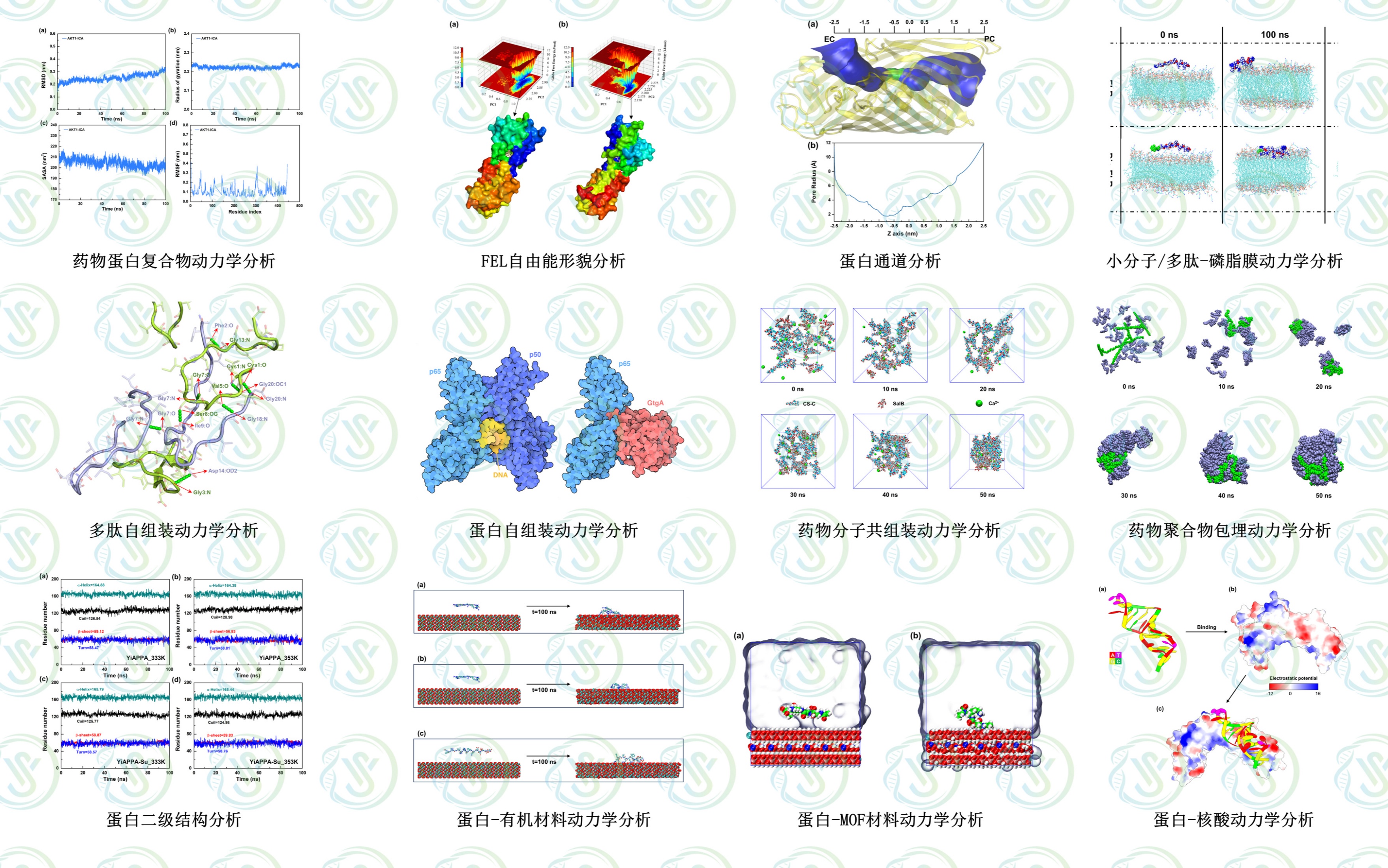
计算体系:生物体系,蛋白质,核酸,多肽,化合物分子,聚合物,有机小分子等
常用软件:Gromacs、Lammps、Amber、MS、Discovery Studio、NAMD等

蛋白三维模型搭建:同源建模、从头建模等
生物三维结构分析:蛋白在不同pH、温度、电场下的三维结构变化等
动力学研究:生物体系的弱相互作用分析、受体-配体组装过程、结合自由能分析、材料体系的高分子构象预测、材料与溶液界面性质、粗粒化模拟等
动力学后数据分析:回旋半径(RMsF)、径向分布函数(RDF)、扩散系数、RMSD、各种能量分析、氢键数量分析、亲疏水性分析等
药物相关模拟:药物衍生物库设计、虚拟筛选、成药性预测、毒性分析、QSAR预测模型构建等

1、有强大的计算资源,可计算ms级时长
2、多年模拟经验积累,能精准算出体系性质,并合理分析
3、提供全面的报告及后续解读
4、超长售后保证,问题实时解答